Published: Mar 27, 2019
合成生物学(synthetic biology)是一门利用工程学设计理念来构件具有不同功能生物体系的新兴学科【1】。近年来,合成生物学在生物能源、生物医药和生命合成等领域取得了长足发展和重要突破,其巨大的潜在价值使其成为各国政府争相抢占的资助领域和各科研机构的研究热点。
全细胞生物传感器(whole-cell biosensor)是以细胞为主要感受元件,可用于检测并报告某种特定物质、环境【2,3】。与传统的化学和电子检测方法相比,它们体积小、易生产、价格低、可降解,因此在环境检测和临床诊断上有极高的应用价值【4-7】。以细胞构建生物传感器已有30多年的历史,但是少有能进入传感器市场。究其原因,可总结为以下三点:1)基因改造生物带来的潜在生物安全问题;2)低效的传感功能;3)缺乏低价易用的传感平台。
近日,英国爱丁堡大学王宝俊研究团队(万心怡为第一作者)在Nature Chemical Biology发表了题为Cascaded amplifying circuits enable ultrasensitive cellular sensors for toxic metals的文章【8】,报道了一套针对全细胞生物传感器的优化方案(图1),可通用于多种全细胞生物传感器,并能大大提高其检测灵敏度和信号输出强度,从而优化传感功能来解决上述第二点问题。


全细胞生物传感器由传感模块(sensing module)、信号处理模块(computing module)和输出/执行模块(output actuating module)构成【2,3,6】。传感模块通常由信号受体蛋白(receptor)和相应的启动子(promoter)组成。该优化方案首先通过调节细胞内受体蛋白的密度来提高传感器的灵敏度,其次通过串联多极转录放大器(transcriptional amplifier)来促进输出模块的信号强度,最后结合转录调节(transcriptional regulation)和翻译后调节(post-translational regulation)的方法来降低在无输入信号时传感器的信号输出(basal expression/leakage),从而增加输出信号的动态范围(output dynamic range)。该优化方案已被用于检测砷和汞的微生物传感器,并增加了其灵敏度高达5,000倍、信号输出强度高达750倍,使其检测限(limit of detection)大大低于砷和汞在饮用水中的安全指标。
此外,为了解决上述第三点问题,该研究利用上述优化方案构建了多种具有不同检测限的微生物传感器,并通过排列这些传感器开发了一种价格低廉且易用的微生物传感器阵列(microbial sensor array)(图1,2)。该传感器阵列可提供类似于音量条的可视化信号输出,并根据不同的信号输入强度提供不同的信号输出图案。该传感阵列还可与多种移动设备(比如USB显微镜和手机)结合来读取输出图案。为了检验该传感阵列,该研究利用5 – 6种砷离子微生物感受器构建了检测砷离子的微生物传感阵列,并用孟加拉国当地有砷污染的地下水试验。结果显示该砷离子传感阵列可准确检测饮用水中1.6 – 175.8 ppb (g/L)的砷离子(图2d),达到并超出了WHO的安全检测指标(10 ppb)。
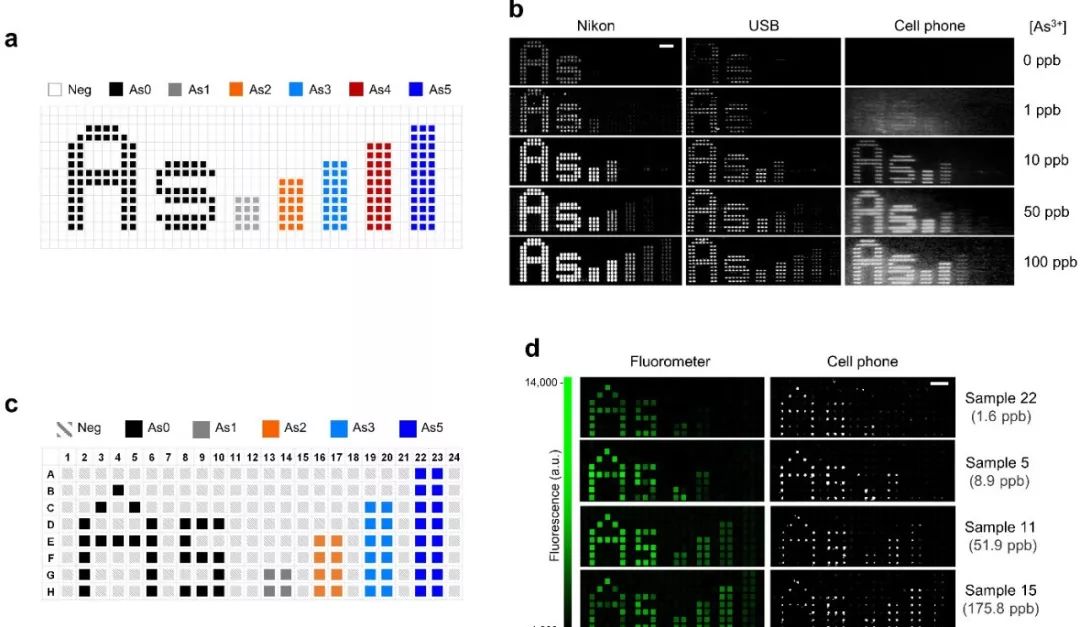
与传统的以单一传感器为基础的传感平台相比,读取多重传感器所提供的信号输出将更为便捷,并更容易被广泛人群接受。不仅如此,该传感器阵列利用水凝胶或微流体装置限制了细胞的流动,从而能够预防GMO传播可能导致的生物安全问题。
本研究提供了一套完整的全细胞传感器优化方案,并构建了低价易用的砷离子传感平台。目前全世界有50多个国家、1.4亿人口的饮用水中有超过10 ppb的砷污染【9】,而孟加拉国每年有4.3 万人死于砷中毒【10】。研究者们希望该砷离子传感平台能够可以为砷污染地区提供低价、便捷的砷污染检测方法。此外,本研究的优化方案与传感平台理念可被广泛用于多种其他细胞体系甚至非细胞体系传感器【11】,希望其能被早日用于环境监测和临床诊断等实际应用中。
原文链接
https://doi.org/10.1038/s41589-019-0244-3
参考文献
-
Bashor, C. J. & Collins, J. J. Understanding Biological Regulation Through Synthetic Biology. Annu. Rev. Biophys. 47, 399–423 (2018).
-
Wang, B. & Buck, M. Customizing cell signaling using engineered genetic logic circuits. Trends Microbiol. 20, 376–384 (2012).
-
van der Meer, J. R. & Belkin, S. Where microbiology meets microengineering: Design and applications of reporter bacteria. Nat. Rev. Microbiol. 8, 511–522 (2010).
-
Ho, C. L. et al. Engineered commensal microbes for diet-mediated colorectal-cancer chemoprevention. Nat. Biomed. Eng. 2, 27–37 (2018).
-
Belkin, S. et al. Remote detection of buried landmines using a bacterial sensor. Nat. Biotechnol. 35, 308–310 (2017).
-
Kim, H. J., Jeong, H. & Lee, S. J. Synthetic biology for microbial heavy metal biosensors. Anal. Bioanal. Chem. 410, 1191–1203 (2018).
-
Mimee, M. et al. An ingestible bacterial-electronic system to monitor gastrointestinal health. Science (80-. ). 360, 915–918 (2018).
-
Wan, X. et al. Cascaded amplifying circuits enable ultrasensitive cellular sensors for toxic metals. Nat. Chem. Biol. in press (2019). doi:https://doi.org/10.1038/s41589-019-0244-3
-
Ravenscroft, P., Brammer, H. & Richards, K. Arsenic Pollution: A Global Synthesis. Arsenic Pollution: A Global Synthesis (2009). doi:10.1002/9781444308785
-
Flanagan, S., Johnston, R. & Zheng, Y. Arsenic in tube well water in Bangladesh: health and economic impacts and implications for arsenic mitigation. Bull. World Health Organ. 90, 839–846 (2012).
-
Pardee, K. et al. Paper-Based Synthetic Gene Networks. Cell 159, 940–954 (2014).

