Published: Aug 27, 2019
CRISPR介导的基因激活(CRISPRa, CRISPR activation)是一类具有巨大应用潜力的基因调控技术,其独特的可编程性和可扩展性理论上能够大幅提高基因激活调控元件的通用性和标准化程度,因此一直是合成生物学领域备受瞩目的研究对象。
尽管CRISPRa在真核细胞中已经相当成熟【1】,但在原核细胞中,少数成功搭建的CRISPRa装置依然受限于其基因激活机制的自身特点【2】。如CRISPR激活复合体的有效靶点范围狭窄,不支持单启动子的多靶点激活;激活输出倍数低,无法支持有效的多通道激活和级联调控。
2019年8月26日,英国爱丁堡大学王宝俊研究团队(刘阳为第一作者)在Nature Communications发表长文Engineered CRISPRa enables programmable eukaryote-like gene activation in bacteria【3】,报道了一种新型“类真核式”CRISPR基因激活系统。该系统巧妙利用了细菌σ54因子独特的转录起始“闭锁”机制,将原核细胞CRISPRa的激活输出倍数提高了一个数量级以上,并首次实现了细菌中CRISPR激活的多通道调控和级联调控。

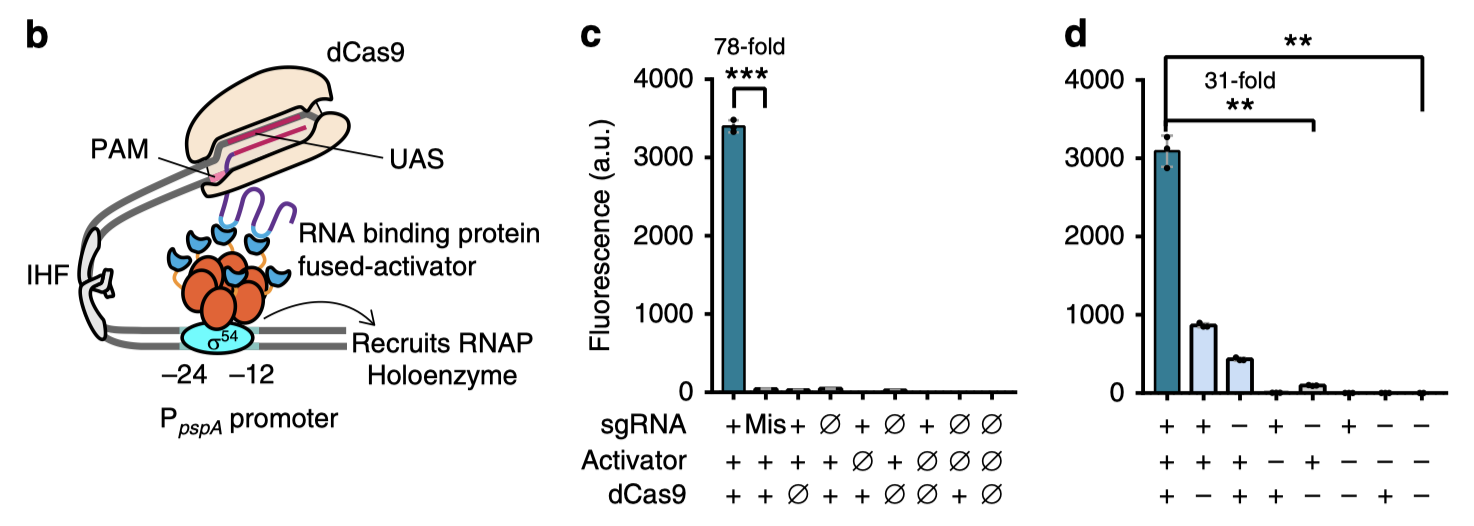
远离启动子核心序列的上游增强子通过DNA成环影响转录起始,进而调控基因表达,这对于熟悉真核生物基因调控的读者并不陌生。与人们熟知的σ70因子依赖型启动子不同,细菌中另一类广泛存在的σ54因子依赖型启动子也有类似机制,这类启动子通常控制环境响应基因的表达。原核细胞许多重要的生理功能如生物固氮、集群效应、致病性、生物膜生成、环境压力耐受等均受这种基因激活机制的控制【4-6】。这些生理功能对基因表达开闭的调节性能要求较高,因此原核生物进化出了这种“类真核”的远程调控机制实现对此类基因的精确控制。
σ54因子依赖型激活子(下文称σ54激活子)是一类具有ATP水解活性的调控蛋白,通常通过组装成六聚体并结合到启动子上游的增强子位点形成一把“分子钥匙”,与结合在启动子核心序列的σ54因子相互作用,解除σ54因子对转录起始的“闭锁”状态。这种激活调控机制具有泄漏表达量低,激活倍率高的特点,曾被用于经典的合成生物学逻辑门装置设计与构建研究【7】。
长久以来,σ54激活子复杂的装配过程和参差不齐的模块性特点是其难以与CRISPR系统结合的主要障碍。王宝俊团队通过结合现有sgRNA支架设计和已知模块性良好的σ54激活子PspF【8,9】,成功实现了σ54因子依赖型CRISPR激活,并展现出了优越的调控性能。研究者进一步全面探索了对σ54激活子的工程化改造及优化策略,并在大肠杆菌和玉米细菌性枯萎病菌(Erwinia stewartii)中发现了另外两种可以与CRISPR/Cas系统结合的激活子。
CRISPR调控系统可编程性的最大价值之一在于其潜在的多通道调控能力,这种可编程的多通道调控必须基于以sgRNA作为单一输入变量,即在保持dCas9和激活子表达水平恒定的情形下,仅通过控制sgRNA的表达量就能有效开闭目标基因。这就要求激活装置对sgRNA泄漏转录具有极高的耐受性。文章作者深入研究了完全基于sgRNA水平单一输入因素的CRISPRa输出特性。通过在sgRNA支架序列中引入突变和利用dCas9的突变体等多种策略降低了整个系统对低浓度sgRNA的敏感性,进而将该CRISPRa系统的激活输出倍数提高到足以支持多通道激活和级联激活的水平上。
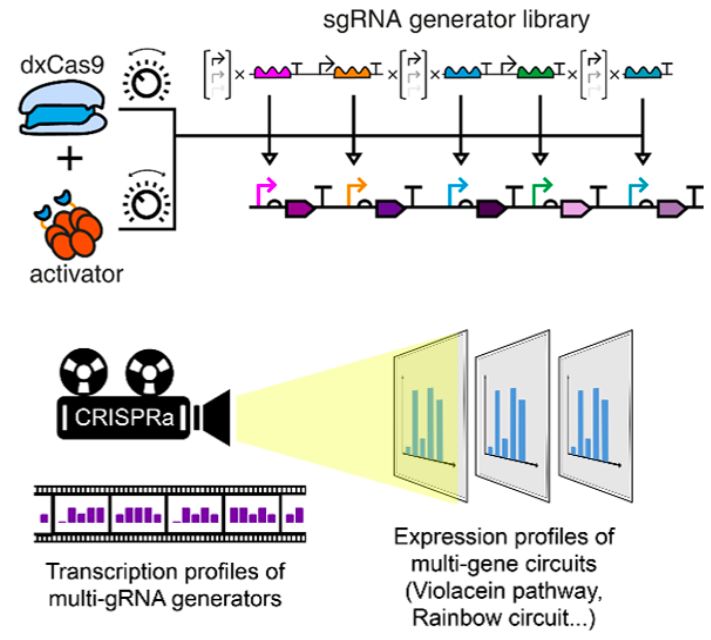
在上述成果基础上,王宝俊团队创造性地提出了一种多基因代谢通路表达谱的筛选平台。不同于常见的代谢通路基因线路的文库构建方法,研究者将多基因表达比例关系的多样性信息从代谢通路线路上分离出来,“预存”在一个通用的多通道sgRNA发生器文库中。通过将sgRNA发生器文库与靶线路配合使用,多样的多sgRNA转录比例关系可以被CRISPRa系统方便地投射到靶标多基因代谢通路线路上,形成多样的靶线路多基因表达量谱,以用于多基因表达比例的筛选和优化(图2)。在这一过程中,投射强度可以被精确控制,从而有效避免待优化线路因潜在毒性或代谢负担所造成的合成或筛选问题。实验结果证明,这一平台具有优良的基因表达控制稳定性。
这一平台首次提出并实现了“可重复使用”多基因表达谱文库的概念。不同的多基因表达线路,只需按照标准化的设计原则进行设计,便可只构建一个单一质粒,再利用规模化生产的通用多sgRNA转录文库“赋予”其各种各样的基因表达比例关系并进行筛选,有望极大降低原核生物多基因表达通路的优化成本,具有广阔的应用前景。
“类似于可回收的火箭,用户不需要再为每一个待优化的多基因线路构建文库——只需要挑选一款通用的sgRNA发生器文库,一次将大量预存的表达谱“投影”到目标线路上就可以了。”
原文链接
https://doi.org/10.1038/s41467-019-11479-0
参考文献
-
Qi, L.S., Larson, M.H., Gilbert, L.A., Doudna, J.A., Weissman, J.S., Arkin, A.P. & Lim, W.A. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell 152, 1173-1183 (2013).
-
Dong, C., Fontana, J., Patel, A., Carothers, J.M. & Zalatan, J.G. Synthetic CRISPR-Cas gene activators for transcriptional reprogramming in bacteria. Nat Commun9:2489 (2018).
-
Liu Y, Wan X, Wang B Engineered CRISPRa enables programmable eukaryote-like gene activation in bacteria, Nat Commun (2019). https://doi.org/10.1038/s41467-019-11479-0
-
Jovanovic, M., James, E.H., Burrows, P.C., Rego, F.G., Buck, M. & Schumacher, J. Regulation of the co-evolved HrpR and HrpS AAA+ proteins required for Pseudomonas syringae pathogenicity. Nat Commun 2:177 (2011).
-
Wolfe, A.J., Millikan, D.S., Campbell, J.M. & Visick, K.L. Vibrio fischeri σ54 controls motility, biofilm formation, luminescence, and colonization. Appl Environ Microbiol 70, 2520-2524 (2004).
-
Charlton, W., Cannon, W. & Buck, M. The Klebsiella Pneumoniae Nifj Promoter - Analysis of Promoter Elements Regulating Activation by the NifA Promoter. Mol Microbiol 7, 1007-1021 (1993).
-
Wang, B., Kitney, R.I., Joly, N. & Buck, M. Engineering modular and orthogonal genetic logic gates for robust digital-like synthetic biology. Nat Commun 2:508 (2011).
-
Zhou, Y., Asahara, H., Schneider, N., Dranchak, P., Inglese, J. & Chong, S. Engineering bacterial transcription regulation to create a synthetic in vitro two-hybrid system for protein interaction assays. J Am Chem Soc 136, 14031-14038 (2014).
-
Ma, H., Tu, L.C., Naseri, A., Huisman, M., Zhang, S., Grunwald, D. & Pederson, T. Multiplexed labeling of genomic loci with dCas9 and engineered sgRNAs using CRISPRainbow. Nat Biotechnol 34, 528-530 (2016).

