Published: Apr 30, 2022
可编程性是CRISPR系统得以发展成为热门分子工具的关键。以CRISPR/Cas9为代表的II型CRISPR系统得到研究和工程化改造较早,从而成为最广为人知的CRISPR类型。遗憾的是,在CRISPR/Cas9系统工程化之初,其双RNA介导所依赖的crRNA与tracrRNA就普遍地被人工融合为单RNA分子(sgRNA)使用,这一分子工具改造的经典范例却亦掩盖了crRNA-tracrRNA配对的可编程性。
2022年4月11日,浙江大学王宝俊研究团队(刘阳博士为第一作者)在Nature Communications 发表长文 Reprogrammed tracrRNAs enable repurposing of RNAs as crRNAs and sequence-specific RNA biosensors,全面揭示了crRNA与tracrRNA配对的可编程性。该研究不仅展示了对crRNA-tracrRNA进行重编程的一般性规律,开发了双gRNA介导的CRISPR激活(CRISPRa)系统。还将小RNA、mRNA、病毒RNA等多种RNA分子劫持为crRNA,实现了利用细菌环境响应基因转录本(含sRNA和mRNA)探测环境污染物,发明了SARS-CoV-2病毒RNA传感器“阿加莎”。该研究曾于去年5月在biorXiv上释出预印本,作为对同行相关研究的呼应和印证。

这项研究源自该研究团队在开发原核CRISPR激活装置的过程中(详见Bioart 2019年报道:Nat Comm|新型原核CRISPRa实现“可投影”多基因表达谱)【1】,注意到sgRNA骨架能够耐受RNA碱基对的改变。研究团队由此猜测crRNA与tracrRNA的配对应具有一定的可编程性。通过将原核CRISPR激活装置改造为双RNA(dual-RNA)介导的系统, crRNA-tracrRNA配对这一令人惊讶的性质被揭露出来(图1)。
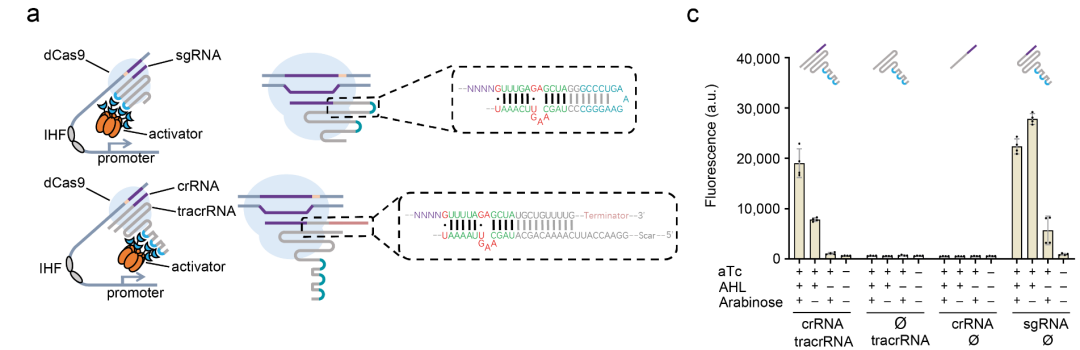
在与清华大学谢震研究团队的后续合作中,研究人员发现,随机成对序列文库中高达70%的重编程crRNA-tracrRNA对呈现出相对野生型(WT)序列50%以上的gRNA活性。基于上述发现,研究团队开发了一套可以通过配对序列变更扩展的“与”门(AND-gate)装置,丰富了合成生物学可编程生物计算元件工具箱(图2)。

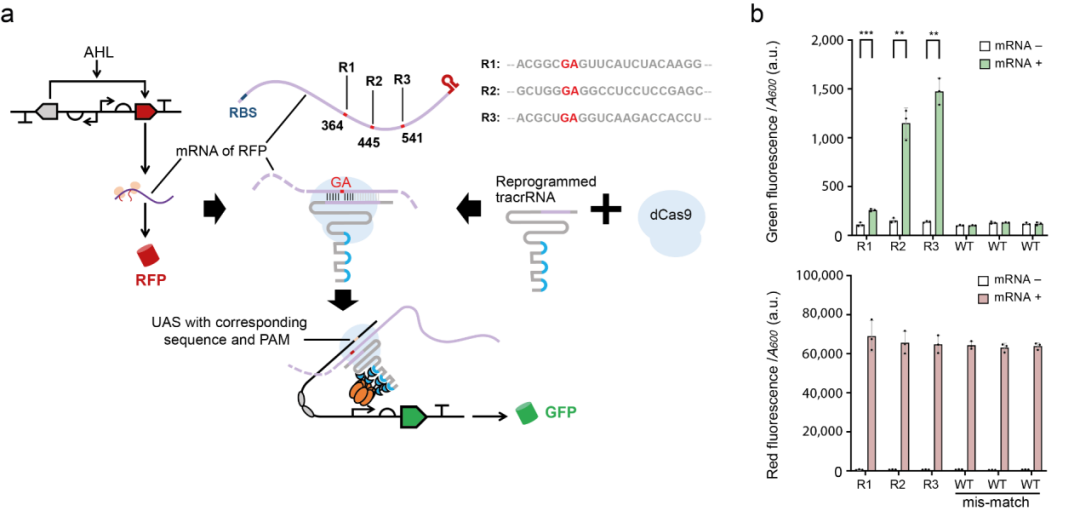
II型CRISPR系统的crRNA结构简单,仅包含与靶DNA配对的spacer段和与tracrRNA配对的repeat段。传统上repeat序列被认为是与相应Cas蛋白共进化的产物【2】,具有保守性。作者大胆推翻上述观念,推测既然crRNA的repeat段序列可变,则意味crRNA没有序列上的限制,反过来,任何序列的RNA分子都有成为crRNA的可能性。
研究团队选用红色荧光蛋白mRNA对上述猜想进行验证。结果表明,RFP mRNA可以作为crRNA参与形成功能性CRISPRa复合体并激活相应启动子(图3)。经过优化,mRNA诱导的基因激活调控倍数可达50倍以上,且CRISPRa复合体对mRNA的翻译过程没有显著干扰。
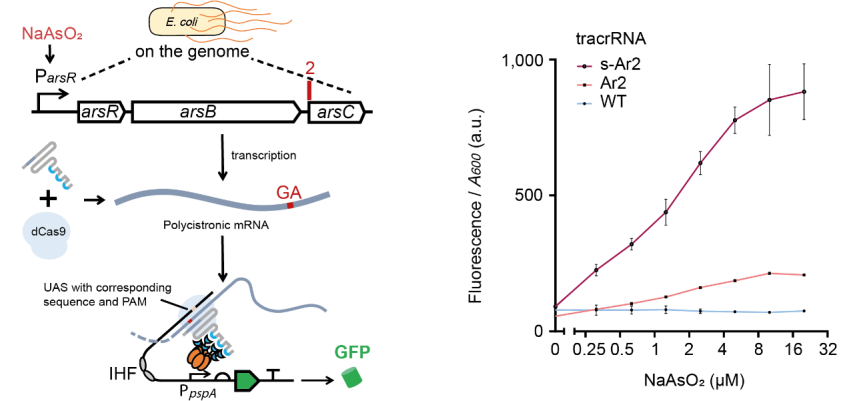
以可编程的方式劫持体内外转录物以操控目标基因,是合成生物学家长期以来梦寐以求的基因网络重构工具。为展示这一工具的功能和通用性,作者劫持了细菌基因组砷响应基因、氧化应激基因、锌/铅离子响应基因、锌/镉响应基因和铜离子响应基因的转录物(含mRNA和sRNA),与人工报告基因线路相连,从而成功探测了培养环境中的砷、过氧化物、锌、铅、铜(图4)。与传统合成生物学全细胞生物传感器相比,该方法不大幅改变宿主细胞中关键感受器蛋白的浓度和靶启动子拷贝数,也不触发宿主细胞中潜在的反馈通路,直接征用细菌在漫长演化过程中形成的鲁棒的生物传感器机制为人类工作。
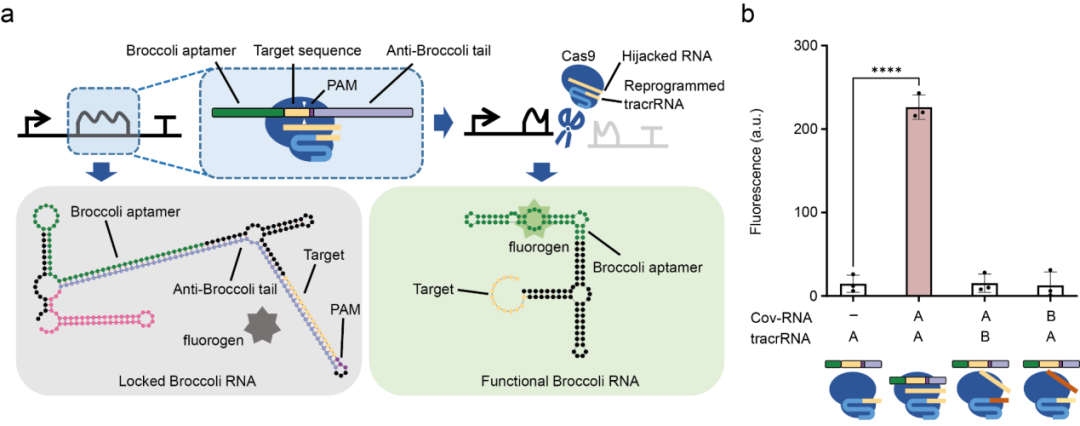
此外,研究团队还利用这一机制发明了新冠病毒核酸检测装置。其原理十分巧妙,在无细胞(cell-free)转录体系中,添加一段编码病毒RNA靶点序列和荧光RNA配体序列的DNA片段。该片段正常转录时,转录物折叠为一个无效二级结构。当体系中出现病毒RNA片段,病毒RNA被劫持为crRNA并参与形成CRISPR复合体,进而切割靶DNA,从被CRISPR截短的DNA源源不断产生的新转录物可以折叠为功能性Broccoli荧光RNA配体,产生荧光信号。该体系可以在添加病毒RNA后的10分钟内快速响应,被设计者命名为非典型gRNA介导的转录终止告警系统:Atypical gRNA-activated Transcription Halting Alarm (AGATHA),(图5)。
“II型CRISPR系统tracrRNA与crRNA互补配对的可编程性是CRISPR/Cas9系统可编程性的最后一片拼图,利用重编程的tracrRNA劫持各种RNA分子激活CRISPR系统,将为编程生物学带来无穷的可能性。” —— 刘阳博士(现就职于剑桥分子生物学实验室LMB)
本项研究获得了众多合作实验室的大力支持和帮助,包括清华大学谢震教授团队,英国爱丁堡大学的Chris French教授团队、克兰菲尔德大学杨竹根博士和格拉斯哥大学Jon Cooper院士团队。
原文链接
https://doi.org/10.1038/s41467-022-29604-x
参考文献
1 Liu, Y., Wan, X. & Wang, B. Engineered CRISPRa enables programmable eukaryote-like gene activation in bacteria. Nature communications 10, 3693, (2019).
2 Chylinski, K., Le Rhun, A. & Charpentier, E. The tracrRNA and Cas9 families of type II CRISPR-Cas immunity systems. RNA biology 10, 726-737, (2013).

